Qu'est-ce que la méthylation de l'ADN ?
La méthylation de l'ADN fait référence à l'ajout d'un groupe méthyle (CH3) à la chaîne d'ADN, catalysé par des ADN méthyltransférases (DNMTs). L'ADN contient quatre types de bases azotées, à savoir la cytosine, la guanine, la thymine et l'adénine (Figure 1A). Les chercheurs ont découvert que la cytosine et l'adénine peuvent être méthylées. La méthylation de la cytosine se produit souvent au niveau de la position 5 du carbone de la cytosine (5-méthylcytosine ou 5mC), qui se trouve exclusivement sur des sites CG symétriques dans la double hélice d'ADN à travers tout le génome, à savoir l'île CpG (Figure 2B). La méthylation de la cytosine est répandue chez les eucaryotes et les procaryotes (Cloney, 2016 ; Cooper, 1983). La méthylation de l'adénine se produit au niveau de la position 6 de l'azote de l'adénine (N6-méthyladénine ou N6mA). La méthylation de l'adénine a été observée dans l'ADN bactérien, végétal et mammifère (Ratel). et al.., 2006 ; Wu et al.., 2016), mais a reçu considérablement moins d'attention (Figure 2B).
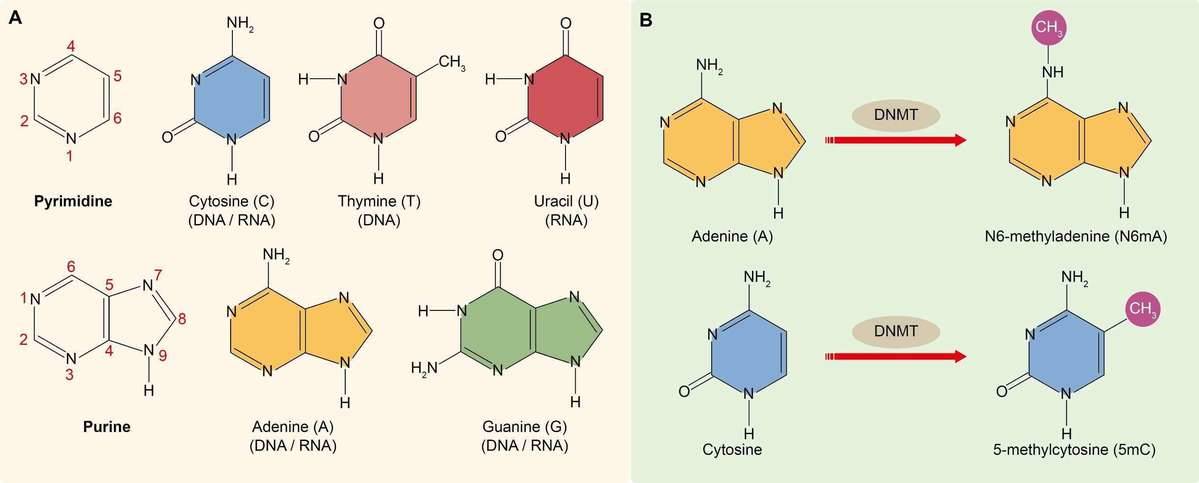 Figure 1. (A) Bases azotées trouvées dans l'ADN et l'ARN. (B) La méthylation de la cytosine et de l'adénine.
Figure 1. (A) Bases azotées trouvées dans l'ADN et l'ARN. (B) La méthylation de la cytosine et de l'adénine.
La méthylation de l'ADN est l'une des modifications épigénétiques les plus importantes, corrélée à la répression génique (Figure 2) et connue pour jouer un rôle essentiel dans le développement embryonnaire, le maintien de la pluripotence, l'imprégnation génomique et l'inactivation du chromosome X à travers la régulation de la transcription, de la structure de la chromatine et de la stabilité des chromosomes (Robertson, 2005). La méthylation de l'ADN peut également affecter la santé, entraînant des cancers, des maladies auto-immunes, des troubles neurologiques ou d'autres maladies. Dans de nombreux processus pathologiques, les îlots CpG des promoteurs géniques acquièrent une hyperméthylation anormale, ce qui entraîne un silence transcriptionnel pouvant être hérité par les cellules filles après la division cellulaire (Wang et Lei, 2018). Les altérations des motifs de méthylation de l'ADN ont été reconnues comme un composant important du développement du cancer (Figure 3).
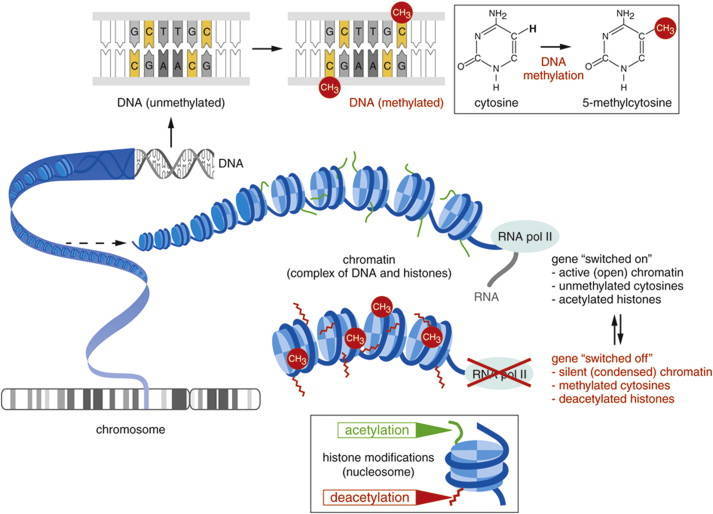 Figure 2. L'expression génique peut être régulée avant le début de la transcription par la modification chimique de l'ADN ou des protéines histones associées (méthylation de l'ADN et acétylation des histones) (Mukherjee et al.., 2015).
Figure 2. L'expression génique peut être régulée avant le début de la transcription par la modification chimique de l'ADN ou des protéines histones associées (méthylation de l'ADN et acétylation des histones) (Mukherjee et al.., 2015).
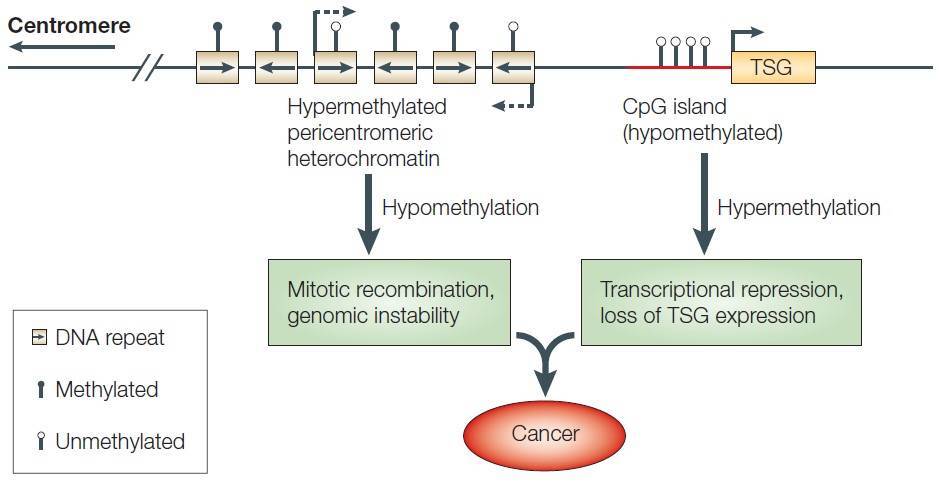 Figure 3. Méthylation de l'ADN et cancer. TSG : gène suppresseur de tumeur. (Robertson, 2005)
Figure 3. Méthylation de l'ADN et cancer. TSG : gène suppresseur de tumeur. (Robertson, 2005)
Détection de la méthylation de l'ADN et RRBS
Comprendre le rôle de la méthylation de l'ADN dans le développement et la maladie nécessite une connaissance de la distribution de ces modifications dans le génome. et al.., 2010). Mesurer la quantité totale de 5mC ou 5hmC (5-hydroxyméthylcytosine) permet aux chercheurs d'obtenir des informations sur des processus biologiques profonds et d'identifier des biomarqueurs pour les maladies. La détection des motifs de méthylation de l'ADN est un domaine de recherche en pleine expansion et plusieurs méthodes sont disponibles pour l'évaluation de la méthylation de l'ADN, le traitement au bisulfite étant une procédure centrale pour la majorité. Un algorithme simple pour choisir une méthode appropriée pour l'analyse de la méthylation de l'ADN est illustré dans la Figure 4 (Kurdyukov et Bullock, 2016).
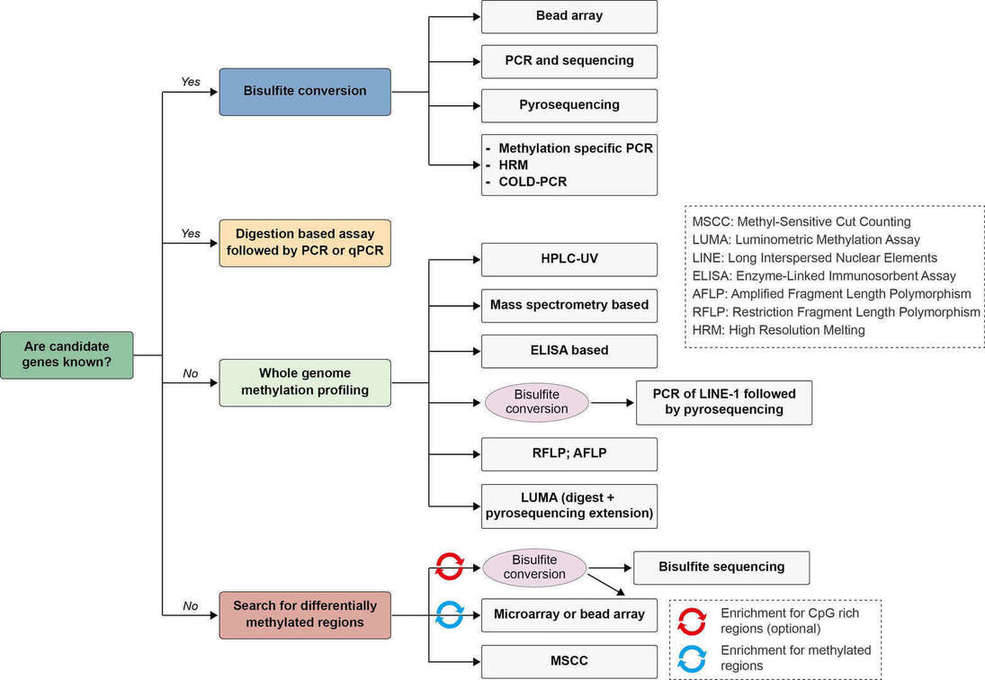 Figure 4. Algorithme pour choisir une méthode appropriée pour l'analyse de la méthylation de l'ADN. Adapté de (Kurdyukov et Bullock, 2016)
Figure 4. Algorithme pour choisir une méthode appropriée pour l'analyse de la méthylation de l'ADN. Adapté de (Kurdyukov et Bullock, 2016)
Le séquençage par bisulfite est l'utilisation d'un traitement au bisulfite de l'ADN pour déterminer son schéma de méthylation. et al.., 1992). Le traitement par bisulfite de l'ADN entraîne la désamination de la cytosine en uracile, et ces résidus convertis seront lus comme de la thymine, comme déterminé par amplification PCR et analyse de séquençage ultérieure (Figure 5).
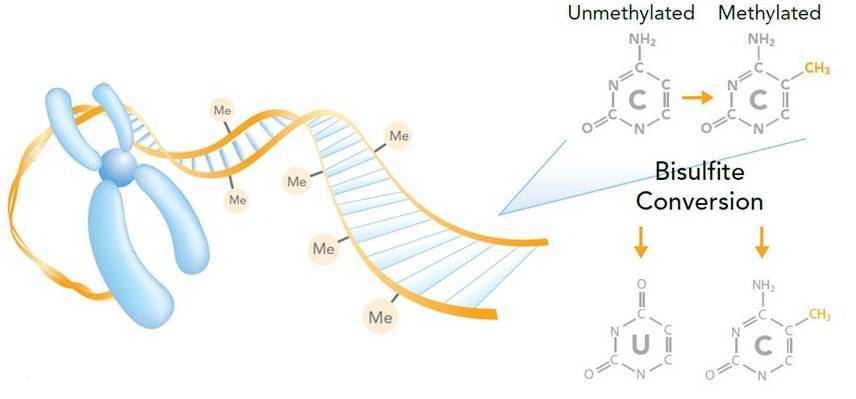 Figure 5. Mesure de la méthylation de l'ADN par séquençage au bisulfite (Imagé par Illumina).
Figure 5. Mesure de la méthylation de l'ADN par séquençage au bisulfite (Imagé par Illumina).
Séquençage bisulfite de tout le génome (WGBS) est la méthode la plus complète d'analyse de la méthylation de l'ADN, et est désormais considérée comme la méthode "de référence" dans les études de méthylation de l'ADN. Les seules limitations sont le coût et les difficultés dans l'analyse des données NGS. Séquençage bisulfite à représentation réduite (RRBS), qui combine des enzymes de restriction et le séquençage au bisulfite pour enrichir les zones du génome avec un contenu élevé en CpG, est une technique efficace et à haut débit pour analyser les profils de méthylation à l'échelle du génome au niveau d'un seul nucléotide. La méthode peut réduire le nombre de nucléotides nécessaires au séquençage à 1 % du génome (Meissner et al.., 2005). En raison de son approche économique et productive, RRBS a été utilisé pour profiler rapidement la méthylation aberrante dans le cancer (Smith et al.., 2009) et états de méthylation dans le développement.
Flux de travail de RRBS
Le protocole de RRBS est illustré dans la Figure 6. Le processus se compose de plusieurs étapes :
- Digestion enzymatique
L'ADN génomique est digéré en fragments d'ADN de tailles variées à l'aide d'une enzyme de restriction insensible à la méthylation. MspI est couramment utilisé.
- Préparation de la bibliothèque
Après la digestion enzymatique, une préparation de bibliothèque, un processus consistant en la réparation des extrémités, l'ajout d'une queue A et la ligation d'adaptateurs de séquençage, est réalisée. La réaction de digestion par MspI entraîne des brins avec des extrémités collantes. La réparation des extrémités est nécessaire pour combler le terminal 3’ des extrémités des brins. L'ajout d'une queue A, dans lequel un adénosine supplémentaire est ajouté aux brins positif et négatif, est nécessaire pour la ligation de l'adaptateur de séquence méthylé à l'étape suivante.
- Sélection de taille
Par la suite, une sélection de taille des fragments résultants est effectuée. Différentes tailles de fragments sont séparées par électrophorèse sur gel et purifiées par excision du gel.
- Conversion au bisulfite
Après la sélection de la taille, la conversion par bisulfite est effectuée, au cours de laquelle la cytosine non méthylée est déaminée en uracile.
- Amplification et purification par PCR
L'ADN converti en bisulfite est ensuite amplifié par PCR avec des amorces qui sont complémentaires aux adaptateurs de séquence. Une étape de purification par PCR est nécessaire avant le séquençage.
- Séquençage
Le séquençage est ensuite réalisé en utilisant la technologie de séquençage de nouvelle génération. Les plateformes Illumina sont les plus couramment utilisées.
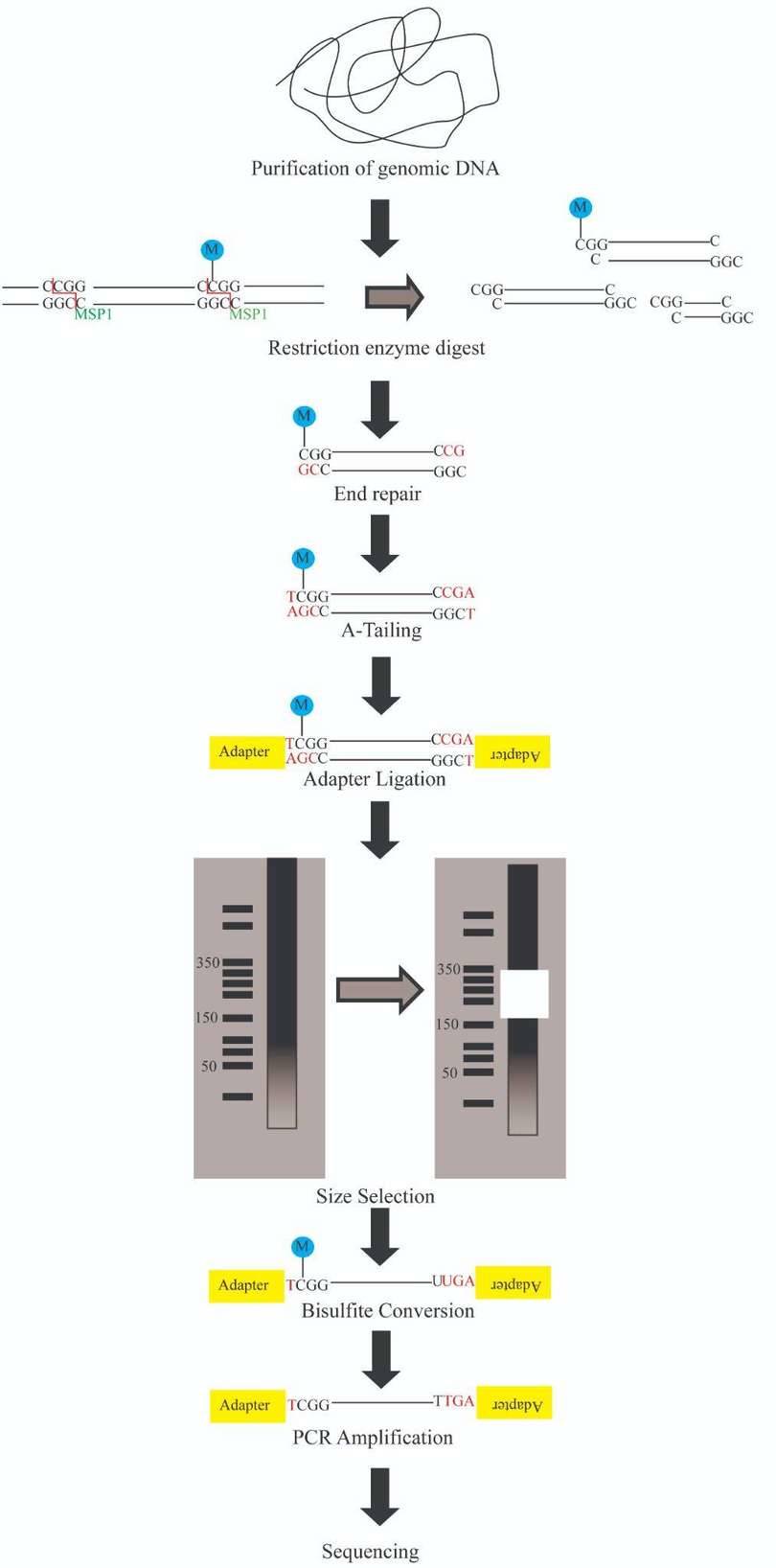 Figure 6. Protocole de séquençage bisulfite à représentation réduite.
Figure 6. Protocole de séquençage bisulfite à représentation réduite.
Avantages et limitations de RRBS
RRBS présente certains avantages techniques par rapport à WGBS et à d'autres méthodes de détection de la méthylation de l'ADN. Mais il existe également certaines limitations dans les étapes spécifiques du protocole.
- Avantages :
- En tirant parti de l'utilisation d'enzymes de restriction spécifiques, le RRBS peut enrichir les régions CpG du génome, réduire la quantité de séquençage nécessaire ainsi que diminuer les coûts.
- Seule une faible concentration d'échantillon est nécessaire pour une analyse de données précise.
- Limitations
- La digestion par les enzymes de restriction ne pouvait pas couvrir toutes les régions CG du génome. Certains CpG sont manqués et certaines régions ont donc une couverture plus faible.
- Des erreurs d'amplification PCR pourraient se produire.
- La conversion complète du bisulfite de l'ADN double brin (ADN db) nécessite une étape de dénaturation, car le séquençage au bisulfite ne convertit que l'ADN simple brin (ADN sb).
Chez CD Genomics, nous sommes dédiés à fournir des services fiables. séquençage épigénomique services, y compris séquençage bisulfite ciblé, séquençage bisulfite à représentation réduite (RRBS), séquençage bisulfite de tout le génome, Séquençage MeDIPet ChIP-seq.
Références :
- Cloney, R. (2016). Méthylation de l'ADN : Une analyse SMRT des épigénomes procaryotes. Nature Reviews Genetics dix-sept, 195.
- Cooper, D.N. (1983). Méthylation de l'ADN eucaryote. Génétique humaine 64, 315-333.
- Frommer, M., McDonald, L.E., Millar, D.S., Collis, C.M., Watt, F., Grigg, G.W., Molloy, P.L., et Paul, C.L. (1992). Un protocole de séquençage génomique qui permet d'obtenir une visualisation positive des résidus de 5-méthylcytosine dans des brins d'ADN individuels. Actes de l'Académie nationale des sciences des États-Unis d'Amérique 89, 1827-1831.
- Harris, R.A., Wang, T., Coarfa, C., Nagarajan, R.P., Hong, C., Downey, S.L., Johnson, B.E., Fouse, S.D., Delaney, A., Zhao, Y., et al. (2010). Comparaison des méthodes basées sur le séquençage pour profiler la méthylation de l'ADN et identification des modifications épigénétiques monoalléliques. Biotechnologie de la nature 28, 1097-1105.
- Kurdyukov, S., et Bullock, M. (2016). Analyse de la méthylation de l'ADN : Choisir la bonne méthode. Biologie 5.
- Meissner, A., Gnirke, A., Bell, G.W., Ramsahoye, B., Lander, E.S., et Jaenisch, R. (2005). Séquençage bisulfite à représentation réduite pour une analyse comparative de la méthylation de l'ADN à haute résolution. Recherche sur les acides nucléiques 33, 5868-5877.
- Mukherjee, K., Twyman, R.M., et Vilcinskas, A. (2015). Les insectes comme modèles pour étudier la base épigénétique des maladies. Progrès en biophysique et biologie moléculaire 118, 69-78.
- Ratel, D., Ravanat, J.L., Berger, F., et Wion, D. (2006). N6-méthyladénine : l'autre base méthylée de l'ADN. BioEssays : actualités et critiques en biologie moléculaire, cellulaire et du développement 28, 309-315.
- Robertson, K.D. (2005). Méthylation de l'ADN et maladies humaines. Revue de la Nature Génétique 6, 597-610.
- Smith, Z.D., Gu, H., Bock, C., Gnirke, A., et Meissner, A. (2009). Séquençage bisulfite à haut débit dans les génomes des mammifères. Méthodes quarante-huit, 226-232.
- Wang, Y.P., et Lei, Q.Y. (2018). Recodage métabolique de l'épigénétique dans le cancer. Communications sur le cancer 38, 25.
- Wu, T.P., Wang, T., Seetin, M.G., Lai, Y., Zhu, S., Lin, K., Liu, Y., Byrum, S.D., Mackintosh, S.G., Zhong, M., et al. (2016). Méthylation de l'ADN sur N(6)-adénine dans les cellules souches embryonnaires mammifères. Nature 532, 329-333.
À des fins de recherche uniquement, non destiné à un diagnostic clinique, un traitement ou des évaluations de santé individuelles.


 Directives de Soumission d'Échantillons
Directives de Soumission d'Échantillons
 Figure 1. (A) Bases azotées trouvées dans l'ADN et l'ARN. (B) La méthylation de la cytosine et de l'adénine.
Figure 1. (A) Bases azotées trouvées dans l'ADN et l'ARN. (B) La méthylation de la cytosine et de l'adénine. Figure 2. L'expression génique peut être régulée avant le début de la transcription par la modification chimique de l'ADN ou des protéines histones associées (méthylation de l'ADN et acétylation des histones) (Mukherjee et al.., 2015).
Figure 2. L'expression génique peut être régulée avant le début de la transcription par la modification chimique de l'ADN ou des protéines histones associées (méthylation de l'ADN et acétylation des histones) (Mukherjee et al.., 2015). Figure 3. Méthylation de l'ADN et cancer. TSG : gène suppresseur de tumeur. (Robertson, 2005)
Figure 3. Méthylation de l'ADN et cancer. TSG : gène suppresseur de tumeur. (Robertson, 2005) Figure 4. Algorithme pour choisir une méthode appropriée pour l'analyse de la méthylation de l'ADN. Adapté de (Kurdyukov et Bullock, 2016)
Figure 4. Algorithme pour choisir une méthode appropriée pour l'analyse de la méthylation de l'ADN. Adapté de (Kurdyukov et Bullock, 2016) Figure 5. Mesure de la méthylation de l'ADN par séquençage au bisulfite (Imagé par Illumina).
Figure 5. Mesure de la méthylation de l'ADN par séquençage au bisulfite (Imagé par Illumina). Figure 6. Protocole de séquençage bisulfite à représentation réduite.
Figure 6. Protocole de séquençage bisulfite à représentation réduite.